Traendo spunto dall’identificazione del nuovo virus che vi abbiamo presentato nel precedente articolo, denominato Hepegivirus-1 umano (HHpgV-1), che coniuga caratteristiche di due generi distinti della famiglia dei Flavivirus, Hepacivirus e Pegivirus, la microbiologa americana Angela Rasmussen commenta in un editoriale gli enormi recenti progressi della diagnostica virologica, che consentono di ipotizzare una significativa accelerazione verso la scoperta di nuovi virus.
Le tecniche di biologia molecolare per l’identificazione dei genomi virali si sono infatti ulteriormente affinate e rese efficienti, fino al sequenziamento ad alta capacità (traduzione un po’ forzata di high-throughput sequencing, HTS), cui si deve la recente scoperta dell’HHpgV-1. Queste tecnologie, a differenza dei metodi PCR classici che sono solo in grado di confermare la presenza di un virus specificamente ricercato, permettono di identificare anche virus il cui genoma è presente in circolo a bassissime concentrazioni, grazie alla eliminazione delle sequenze dell’ospite, ad alta concentrazione, che rappresentano un fattore di disturbo.
Una piattaforma ancora più efficiente in questa direzione risulta la Vir-Cap-Seq-vert (virome-capture-sequencing platform for vertebrate virus), che utilizza una libreria di oligonucleotidi in grado di identificare fino al 98% delle sequenze dei virus noti infettivi per i vertebrati e, pertanto, utile per ‘catturare’ eventuali nuovi genomi virali che abbiano analogie di sequenza con tali virus. Questa tecnica potrà ad esempio essere utilizzata per verificare l’effettiva prevalenza del nuovo HHpgV-1 e studiare sua associazioni con patologie. Si apre dunque una nuova era per l’esplorazione del mondo dei virus.
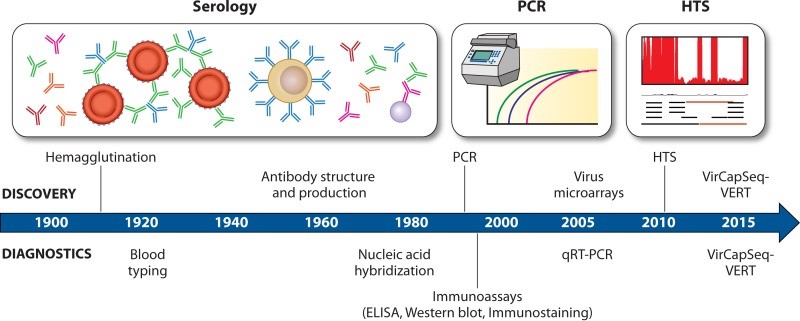
Lo sviluppo delle tecnologie per la diagnostica virologica: dai tests sierologici all’era della PCR negli anni ’90, fino alle più moderne tecniche di sequenziamento dei nostri giorni
Rasmussen AL.
Probing the Viromic Frontiers.
MBio. 2015 Nov 10;6(6):e01767-15.



